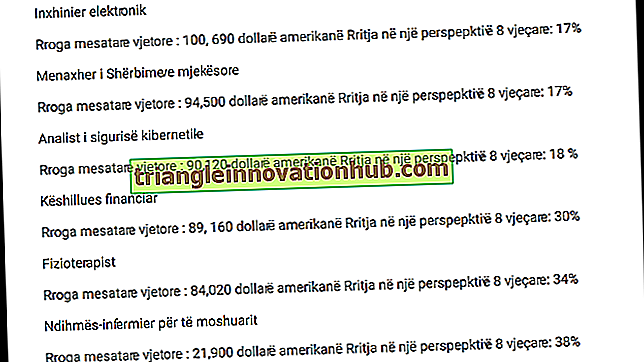
2 Grandes Passos Envolvidos no Mecanismo da Síntese Proteica: Transcrição e Tradução
Os principais passos envolvidos no mecanismo da Síntese de Proteínas são: 1. Transcrição e 2. Tradução!
A biossíntese de proteínas está sob controle direto do DNA na maioria dos casos ou sob o controle do RNA genético onde o DNA está ausente.
A informação para a estrutura de um polipéptido é armazenada numa cadeia polinucleotídica. Em 1958, Crick propôs que a informação presente no DNA (na forma de sequência de base) é transferida para o RNA e depois do RNA é transferida para a proteína (na forma de sequência de aminoácidos), e que essa informação não flui no direção inversa, isto é, de proteína para RNA para DNA.
Moléculas de DNA fornecem as informações para sua própria replicação. Essa relação entre DNA, RNA e moléculas de proteína é conhecida como dogma central. Temin (1970) relatou que os retrovírus operam um dogma central reverso (fluxo inverso de informação) ou feminismo dentro das células hospedeiras.
O RNA genômico desses vírus primeiro sintetiza o DNA através da transcrição reversa; este processo é catalisado pela enzima transcriptase reversa, o ADN transfere depois informação para o ARN mensageiro que toma parte na tradução da informação codificada para formar o polipéptido.

Mecanismo de Síntese Proteica:
(i) Duas etapas principais estão envolvidas na síntese de proteínas; (i) transcrição, envolvendo transferência de informação genética de DNA para mRNA, e (ii) tradução, envolvendo tradução da linguagem de ácido nucléico para a de proteínas.
I. Transcrição:
A transferência de informação genética do DNA para o mRNA é conhecida como transcrição. Uma única RNA polimerase compromete a síntese de todos os RNAs (incluindo mRNA, rRNA e tRNA) em bactérias. Os eucariotos, por outro lado, contêm pelo menos três RNA polimerases distintas.
Um deles localizado no nucléolo é designado por RNA polimerase I ou 'A' e é responsável pela síntese de rRNA. A segunda RNA polimerase eucariótica é encontrada no nucleoplasma, é designada como RNA polimerase II ou 'B' e é responsável pela síntese de precursores de mRNA chamados RNA nuclear heterogêneo (HnRNA).
A terceira RNA polimerase eucariótica também é encontrada no nucleoplasma e é chamada RNA polimerase III ou 'C', que é responsável pela síntese do RNA 5S e do tRNA. Os eucariotos também contêm outras RNA polimerases em mitocôndrias e plastídios.
A RNA polimerase bacteriana consiste em quatro cadeias polipeptídicas diferentes: enzima central x (duas cadeias de ∞ e uma cadeia única de β 'e β) e fator sigma (a)
1. Transcrição do mRNA do DNA:
Na presença de enzima RNA polimerase dependente de DNA, a mensagem genética codificada no DNA é transcrita em mRNA. As duas cadeias da molécula de DNA específica desenrolam-se e uma destas duas cadeias, actua como molde (esta cadeia é chamada cadeia anti-sentido), da qual a sequência exacta de nucleótidos é transcrita na molécula de ARNm. Como resultado, a sequência de bases da molécula de ARNm é complementar à da cadeia anti-sentido que serviu como modelo. Como a síntese de DNA, a síntese de RNA também procede de 5 ′ a 3 ′ direção (5 ′ - 3 3 ′).
(a) Transcrição em procariontes:
Em bactérias, apenas uma única RNA polimerase catalisa a síntese de diferentes tipos de RNAs. A RNA polimerase consiste em quatro cadeias polipeptídicas (αββ'α 2 ) que constituem a enzima central e um fator sigma (σ), que é fracamente ligado à enzima do núcleo. O fator sigma ajuda no reconhecimento de sinais iniciais na molécula de DNA e direciona a RNA polimerase na seleção do local de iniciação. Na ausência de σ, a enzima central inicia a síntese de RNA de maneira aleatória.
Uma vez iniciada a síntese de RNA, uma dissociação e uma enzima central provocam o alongamento da mRNAA.
O mecanismo de transcrição em procariotas envolve, portanto, as seguintes etapas:
1. Ligação de holoenzima de RNA polimerase a um sítio promotor. Um grande número desses sites, principalmente antes do ponto de partida (ou seja, a montante), mas raramente também após o ponto inicial (isto é, a jusante), foram identificados.
2. Desenrolamento do DNA, levando à separação de dois filamentos, dos quais apenas um é transcrito.
3. Dissociação do fator sigma (a).
4. Elongação do transcrito de mRNA com a ajuda da enzima do núcleo.
5. A terminação da síntese de mRNA é causada pelo códon de terminação no DNA. Em bactérias, este sinal de terminação é reconhecido pelo fator rho (P).

(b) Transcrição de mRNA em eucariotos:
Em eucariotos existem pelo menos dois tipos de RNA polimerases. RNA polimerase-A é responsável pela síntese de rRNA. A RNA polimerase-B produz a síntese de RNA-Hn (RNA nuclear heterogêneo, a partir do DNA. Uma sequência de cerca de 200 nucleotídeos de ácido adenílico - poly A (ácido poliadenílico) se liga à extremidade 3 'do RNA-Hn. Simultaneamente o RNA-Hn se desintegra no final 5. O produto final é conhecido como poli-A-mRNA.
Difunde-se do núcleo para o citoplasma, onde é utilizado para a síntese proteica (ambas as extremidades possuem uma sequência nucleotídica específica. A extremidade 5 'da molécula de mRNA possui 7-metil guanosina, enquanto a extremidade 3' termina na sequência poli A. A seqüência de nucleotídeos nas duas extremidades de todas as moléculas de mRNA é a mesma, portanto, as moléculas de mRNA são chamadas de extremidades marcadas.
Formação de aminoacil-tRNA:
Estudos realizados por Lipmann e colegas de trabalho durante a década de 1950 mostraram que os aminoácidos se ligam às moléculas de tRNA: essa ligação tem as duas etapas a seguir:
1. O primeiro passo consiste na ativação de aminoácidos; uma molécula de aminoácido reage com uma molécula de ATP (trifosfato de adenosina) para produzir uma molécula aminoacil-AMP (amino-acil adenilato) e uma molécula de prifosfato (PP).

2. No segundo passo, o aminoido da molula de aminoacil-AMP transferido para uma molula de ARNt especica e a molula de AMP (monofosfato de adenosina) libertada.

Ambas as reações são catalisadas pela mesma enzima, aminoacil-RNAt sintetase. O complexo aminoácido-AMP está fortemente ligado à enzima durante toda a reação. O grupo carboxilo do aminoido reage com o grupo -OH do resuo fosfato de AMP para formar aminoacil adenilatos, enquanto se liga a um dos grupos -OH da ribose do nucleido de adenina terminal para produzir aminoacil-ARNt.
Cada aminoácido tem sua própria aminoacil-RNAt sintetase distinta, e alguns aminoácidos podem ter mais de uma enzima ativadora.
II. Tradução:
A etapa de tradução envolve a tradução da linguagem de ácidos nucléicos (disponível na forma de mRNA) para a linguagem das proteínas.
O processo de tradução pode ser dividido nas seguintes etapas distintas:
(1) iniciação
(2) alongamento e
(3) rescisão.
1. Iniciação da cadeia polipeptídica:
O início da cadeia polipeptídica é sempre provocado pelo aminoácido metionina, que é codificado pelo códon AUG. Em E. coli, dois tRNAs diferentes recebem metionina - (i) tRNA m met (tRNA nãoformulável) e (ii) tRNA f met (tRNA formilável). ARNt fmm deposita metionina formilada como o primeiro aminoácido da cadeia polipeptídica e inicia assim a formação da cadeia polipeptídica. O ARNt m encontrou depósitos de metionina na posição intercalar na cadeia polipeptídica.
Significa que toda mensagem começa com o códon AUG.
(i) Iniciação da cadeia polipeptídica nos procariontes:
Em procariotas, a iniciação é provocada pela metionina formilada.
Na metionina formilada em E. coli é captada por um tRNA diferente que é designado por tRNA f met (tRNA formilável). A metionina na posi�o intercalar na cadeia polipept�ica �depositada por outro ARNt-ARNt encontrado (ARNt n�form�el).

A metionina formulada se liga ao tRNA f met formando f-met-tRNA f met . A subunidade pequena do ribossoma (30S) liga-se à extremidade 5 'do ARNm que transporta o codão AUG para formar um complexo de iniciação (30S-ARNm). Isto é facilitado por um factor de proteína de iniciação 1F3 f-met-ARNt f met ligado ao complexo de iniciação formando 30S-ARNm-f-met-ARNt de met ; O fator de iniciação 1F 2 é essencial para esta etapa. Isto combina com a grande subunidade (50S) completando a formação dos ribossomos 70S. Esta associação utiliza energia devido à clivagem de uma molécula de GTP.

2. Alongamento da Cadeia Polipeptídica:
Após a formação do 70S-mRNAf-met-ARNt para o complexo encontrado, o alongamento da cadeia polipeptídica ocorre pela adição regular de aminoácidos nas seguintes etapas:
(i) Liga�o de AA-ARNt no s�io-A da subunidade maior do ribossoma:
Muito provavelmente, o complexo aminoacil ARNt (AA-ARNt / - Met-ARNtt é ligado ao local aceitador na subunidade maior do ribossoma (local A) e a cadeia peptídica transportadora de ARNt é ligada ao seu local peptídico ou doador (P Este processo envolve uma molécula de GTP que fornece a energia necessária O segundo aminoacil ARNt liga-se ao sítio A e liga-se ao segundo codão GCU no ARNm Local A para fixação de ARNt.

(ii) Formação de ligação peptídica:
Uma ligação peptídica é formada entre o grupo COOH do peptidil-tRNA no sítio-P e o grupo-NH2 do aminoacil-RNAt do sítio-A. Após a formação da ligação peptídica, o ARNt do sítio P é libertado e a cadeia polipeptídica é transferida para o ARNt presente no local A.
(iii) Movimentação de peptidyl tRNA do sítio A para o sítio P:
Assim que o ARNt do local P é libertado, o peptidil-ARNt do local A regressa ao local P. O processo é completado com o auxílio de uma molécula de GTP e fator de transferência ou enzima translocase.
Durante este processo, o ribossoma desloca-se ao longo do ARNm na direco de 5-3 'de modo a que o cod seguinte no ARNm esteja disponel no local A. Isso requer G-factor e GTP.
À medida que um ribossoma se move ao longo do comprimento do mRNA, o ponto de iniciação no mRNA fica livre. Pode formar um complexo de iniciação com subunidade 30S de outro ribossomo. Deste modo, vários ribossomas são ligados a uma única molécula de ARNm. Este complexo é conhecido como complexo de polirribossomo.
Durante o processo de stese proteica, pode-se observar um certo nero de ribossomas ligados a uma ica molula de ARNm de cada vez, cada um com uma cadeia polipeptica em formao, sendo o tamanho das cadeias polipepticas em diferentes ribossomas diferentes.
Há ainda outro fator de iniciação IF1; que é o menor dos três fatores de iniciação (IF1-9500 daltons, IF2 - 73.000 daltons; IF3 = 23.000 daltons) e cujo papel não é claramente entendido. Pode estar preocupado com a ajuda na liberação de IF2 do complexo de iniciação.
(iv) Iniciação da cadeia polipeptídica em eucariotos:
Em eucariotas, a iniciação da cadeia polipeptídica é provocada por um met-ARNt especial, mas a metionina não é formilada (porque o ARNt de met está ausente nas plantas e a enzima transformalise está ausente em animais). Em eucariotos, unidade menor (40 S) de ribossomo se associa com tRNA iniciador conhecido como tRNAy f .
40S + Met-tRNA eu conheci 40S-Met-tRNA eu conheci
40S - Met-ARNt + ARNm -> 40S-ARNm-metRNA encontrado
40S - mRNA-met-tRNA eu conheci + 60S -> 80S -mRNA-metRNA eu conheci
Em eucariotos existem pelo menos dez fatores de iniciação diferentes. Estes são elF1, eIF2, eIF3, eIF4A, eIF4B, eIF4C, eIF4D, eIF4F, eIF5 e eIF6. eIF3 e eIF2 são análogos a IF2 e IF3 de procariotas.
3. Terminação do Polipeptídeo:
É provocado pela presença de qualquer um dos três codões de terminação, nomeadamente UAA, UAG e UGA. Esses códons de terminação são reconhecidos por um dos dois fatores de liberação RF1 e RF2 em E.coli. Destes fatores de liberação, RF1 reconhece UAA e UAG, enquanto RF2 reconhece UGA e UAA. Eles ajudam o ribossomo a reconhecer esses trigêmeos.
Os fatores de liberação parecem atuar em A 'já que o tRNA supressor capaz de reconhecer códons de terminação pode competir com fatores de liberação por entrada no local A'. Um terceiro fator de liberação RF3 parece estimular a ação de RF1 e RF2.
Para a reação de liberação, o polipeptidil-tRNA deve estar presente no sítio 'P' e os fatores de liberação auxiliam na divisão do grupo carboxila entre o polipeptídeo e o último tRNA portador dessa cadeia. O polipéptido é assim libertado e o ribossoma dissocia-se em duas subunidades com a ajuda de IF-3.
Em eucariota, apenas um fator de liberação é conhecido, ou seja, eRF1.
4. Modificação da Cadeia Polipeptídica Liberada:
O grupo formilo do primeiro aminoácido, metionina, da cadeia polipeptídica libertada é removido pela enzima deformilase. Algumas outras enzimas, como as expeptidases, removem alguns dos aminoácidos da extremidade do terminal N ou da extremidade do terminal C ou de ambas as extremidades da cadeia polipeptídica. Finalmente esta cadeia polipeptídica sozinha ou junto com outras cadeias é dobrada para assumir a estrutura terciária ou quaternária e se modifica em enzima funcional.